Un algoritmo cuántico probado en dispositivos cuánticos comerciales podría ayudar a descubrir fármacos

PASQAL implementó con éxito por primera vez un algoritmo en un dispositivo cuántico de átomo neutro comercial para abordar un problema crítico de biología molecular en el descubrimiento de fármacos. Este proyecto se realizó en colaboración con Qubit Pharmaceuticals.
Las proteínas son moléculas grandes que desempeñan funciones críticas en nuestro cuerpo, como ayudar a construir y reparar tejidos, impulsar reacciones metabólicas y mantener el equilibrio de líquidos. Sin embargo, debido a que las proteínas son responsables de procesos tan esenciales, también pueden ser dañinas. Si algo sale mal mientras se construye una proteína en una célula, el resultado puede ser una proteína tóxica. Por ejemplo, los cánceres están relacionados con modificaciones que se producen a nivel del ADN, que producen proteínas tóxicas y modifican la capacidad de las células para funcionar con normalidad. De hecho, las proteínas defectuosas son responsables de muchas de las enfermedades del mundo.
Los medicamentos que pueden ayudar a combatir enfermedades suelen ser moléculas más simples y más pequeñas, llamadas ligandos, diseñadas para adherirse a un lugar estratégico de la proteína tóxica para inhibir su acción dañina.
Para diseñar un fármaco, los investigadores deben investigar el proceso de unión entre el fármaco y la proteína dañada objetivo. Debido a que las proteínas viven y funcionan dentro de las células, el proceso de unión entre fármaco y proteína debe entenderse en un ambiente lleno de la sustancia más común en la célula: agua. De hecho, el agua puede tener un impacto dramático en la forma en que una proteína objetivo interactúa con un fármaco.
Búsqueda de moléculas de agua dentro de las proteínas.
Las proteínas son largas cadenas de moléculas llamadas aminoácidos que interactúan entre sí, haciendo que la cadena se doble -como una cinta- y cree estructuras complejas que pueden tener bolsas. El agua de la célula penetra en el interior de la proteína, llena sus bolsas, afecta el tamaño y la estructura de la proteína y media en la interacción entre la proteína y el ligando. Investigar dónde y cuántas moléculas de agua puede tener una proteína en sus bolsillos es crucial para diseñar fármacos capaces de inhibir el comportamiento tóxico de la proteína diana.

Los métodos computacionales para comprender la hidratación de proteínas han avanzado significativamente junto con los enfoques experimentales, ahorrando tiempo y reduciendo costos, facilitando los procesos de descubrimiento de fármacos. Además, en muchos casos, las técnicas computacionales pueden proporcionar una mejor comprensión de la estructura y dinámica del sistema que las configuraciones experimentales. La razón es que las áreas abarrotadas y las cavidades interiores de una proteína requieren una resolución muy alta, mientras que los experimentos cristalográficos de rayos X sólo pueden proporcionar información limitada debido a la mala resolución.
Las técnicas de cálculo numérico pueden resultar muy útiles. Se pueden utilizar para simular la presencia explícita y la interacción de moléculas de agua en una proteína. Sin embargo, estas simulaciones son generalmente costosas y el tiempo necesario para proporcionar predicciones precisas puede ser extremadamente largo, principalmente si la cavidad bajo investigación está suficientemente ocluida.
Un enfoque alternativo es encontrar primero la densidad del agua en las bolsas de proteínas y luego extraer la posición de las moléculas de agua de la densidad.
Sin embargo, el número de configuraciones (las diferentes formas en que se pueden embolsar las moléculas de agua) correspondientes a una densidad determinada sigue siendo potencialmente muy grande para los métodos clásicos.
Un algoritmo híbrido cuántico/clásico para encontrar moléculas de agua dentro de bolsas de proteínas
La computación cuántica es hoy una gran fuente de esperanza para abordar problemas demasiado complejos para las computadoras clásicas. Al explotar los principios de la física cuántica, las unidades de procesamiento cuántico pueden almacenar grandes cantidades de información simultáneamente y funcionar excepcionalmente bien para abordar problemas con una gran cantidad de combinaciones. Vea ejemplos en blogs anteriores aquí y aquí.
PASQAL, en colaboración con Qubit Pharmaceuticals, está desarrollando un enfoque híbrido cuántico/clásico que utiliza un algoritmo clásico para encontrar información sobre la densidad del agua en la proteína y luego un algoritmo cuántico para localizar las moléculas de agua dentro de cualquier bolsillo, incluso en los enterrados. . La colaboración implementará este algoritmo cuántico/clásico de colocación de agua en un dispositivo cuántico PASQAL.
Prueba experimental en Fresnel, unidad comercial de procesamiento cuántico PASQAL
Los científicos de PASQAL han probado con éxito una versión preliminar del nuevo algoritmo cuántico de colocación de agua en Fresnel 1, la primera computadora cuántica industrial de átomo neutro de PASQAL. Para esta prueba de concepto, los científicos utilizaron Major UrinaryProtein-1 (MUP-1), una proteína producida principalmente por el hígado. La evidencia demuestra que esta proteína juega un papel clave en la comunicación a través de feromonas y en la regulación del gasto energético y el metabolismo.

La siguiente figura muestra que los resultados experimentales (a la derecha) concuerdan con la emulación de predicciones teóricas (a la izquierda). Cada figura representa una porción 2D en la bolsa de proteínas donde el sombreado amarillo representa la ubicación del agua.
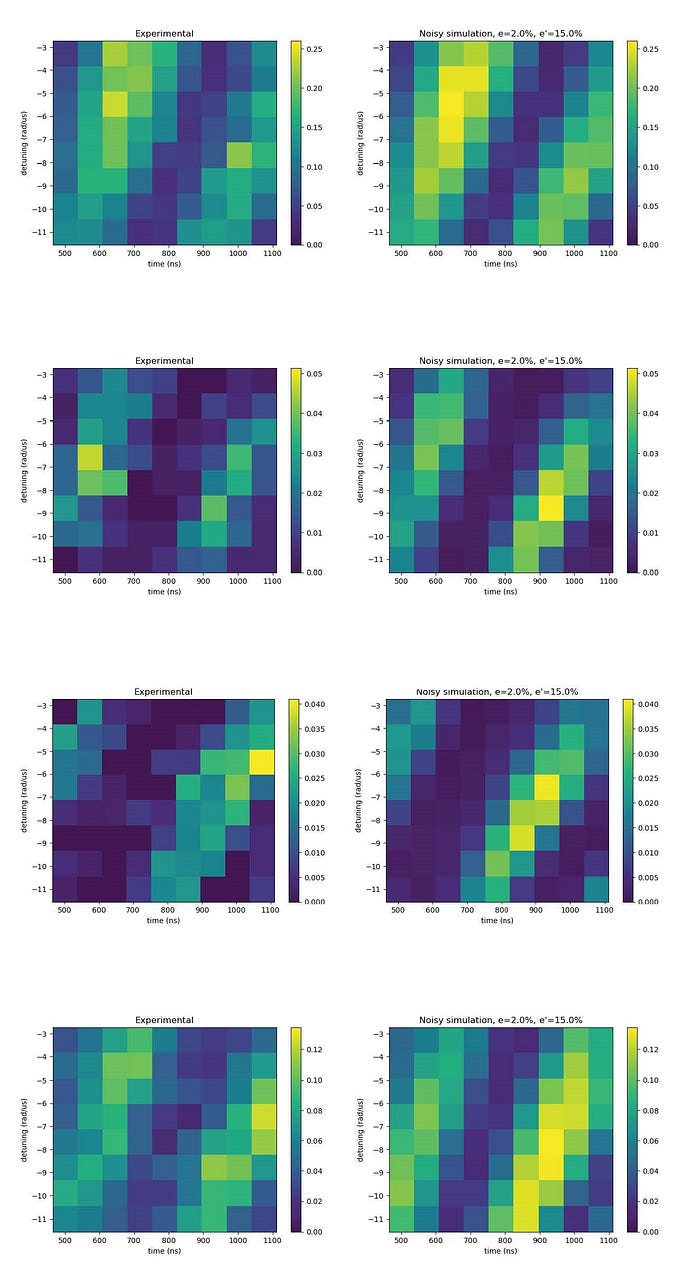
Este resultado exitoso indica que se pueden obtener soluciones significativas al problema de la colocación del agua utilizando dispositivos de átomos neutros, como los de PASQAL. Recientemente se ha publicado una preimpresión con estos resultados en arxiv. Esta es la primera vez que se lleva a cabo un experimento de este tipo utilizando una computadora cuántica real, lo que demuestra la capacidad de las tecnologías cuánticas para contribuir al avance de los problemas de atención médica. La versión completa del algoritmo de colocación de agua PASQAL se implementará en la máquina de átomo neutro de próxima generación que funcionará con 1000 qubits.
Explicación de un algoritmo cuántico para acelerar el descubrimiento de fármacos
El algoritmo cuántico PASQAL utiliza la densidad del agua dentro de las bolsas de proteínas, que se calcula con una computadora clásica utilizando un método llamado Modelo de sitio de interacción de referencia 3D (3D-RISM). El algoritmo 3D-RISM produce una distribución de probabilidad de densidades, que es una especie de patrón continuo y sin estructura, como manchas borrosas en una hoja de papel. La pregunta es, ¿cómo extraemos la posición de cada molécula de agua de ese resultado confuso?
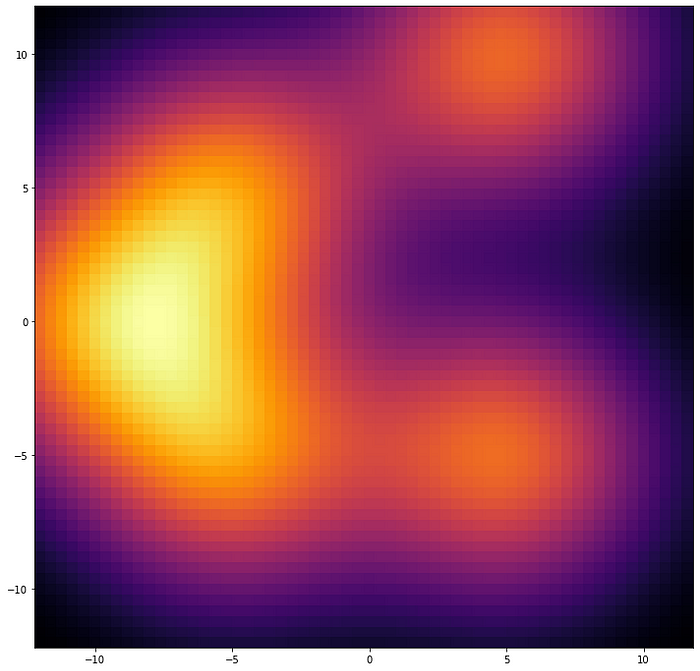
Para esta tarea, PASQAL y Qubit Pharmaceuticals han creado un algoritmo cuántico que partirá de la pregunta contraria: si proponemos una distribución de aguas, ¿reproducirá el mapa de densidad dado por el Modelo 3D de Sitio de Interacción de Referencia?
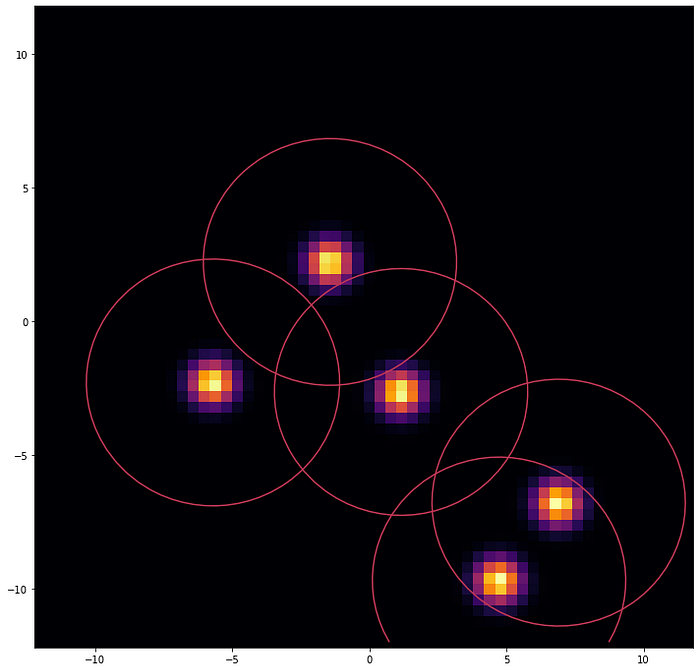
El primer paso es adivinar las posiciones de las moléculas de agua suponiendo lugares de alta densidad de agua, cada posición con una distribución de probabilidad. En otras palabras, los «difuminamos» para que parezcan una distribución continua, difusa y extendida. Luego compare estas configuraciones borrosas con el resultado proporcionado por el modelo de sitio de interacción de referencia 3D utilizando una cantidad que represente la «distancia». La idea es producir tantas combinaciones como sea posible para encontrar la «distancia» mínima entre estos dos patrones de densidad.
Para reducir esta distancia utilizamos el hecho de que, en la vida real, las moléculas de agua están interactuando creando redes a través de enlaces de hidrógeno, y que existe una distancia física mínima entre ellas.
El método cuántico PASQAL está inspirado en el mejor software clásico creado para este trabajo llamado Gasol. Sin embargo, Gasol asigna una penalización incluida artificialmente a las configuraciones en las que dos moléculas de agua se colocan demasiado cerca una de la otra. Esta restricción se implementa naturalmente con arquitecturas de átomos neutros, veamos cómo.
Los dispositivos cuánticos de átomos neutros utilizan láseres altamente enfocados, las llamadas pinzas ópticas, para capturar y manipular átomos neutros individualmente para crear matrices 1D, 2D y 3D en configuraciones arbitrarias. En estas arquitecturas, cada qubit está representado por un estado de energía atómica en dos niveles, normalmente un estado fundamental y un estado de Rydberg, un estado de muy alta energía. En el estado de Rydberg, los átomos están polarizados, lo que induce interacciones de Van der Waals entre ellos.
Esta interacción entre átomos de tipo Van der Waals prohíbe que dos átomos estén en el estado de Rydberg simultáneamente, si están demasiado cerca uno del otro, un fenómeno llamado bloqueo de Rydberg.
En el experimento realizado en Fresnel 1, utilizamos matrices 2D de átomos de rubidio en el estado de Rydberg para representar la posición y distribución de probabilidad de las moléculas de agua dentro de una bolsa de proteínas, utilizando el bloqueo de Rydberg como una forma natural de mantener las aguas demasiado cerca entre sí. otro. otros durante la simulación.
El futuro del diseño cuántico asistido por computadora
Gran parte del esfuerzo de un proyecto de descubrimiento de fármacos se centra en identificar y estudiar el bolsillo para encontrar y optimizar un fármaco con alta afinidad por la proteína objetivo.
A pesar de varios éxitos, sigue siendo extraordinariamente difícil predecir la afinidad de unión entre una proteína y un ligando. La capacidad de diseñar nuevos ligandos según la voluntad de inhibir la acción de biomoléculas nocivas sigue siendo uno de los principales desafíos de las ciencias de la salud contemporáneas. La implementación exitosa de un algoritmo cuántico que ayuda a abordar la afinidad de los ligandos de las proteínas representa un enorme paso adelante hacia la mejora de nuestra calidad de vida.
Referencias
D’Arcangelo, M., Loco, D., equipo de Fresnel, et al. (2023). La computación cuántica analógica aprovecha los átomos neutros para la predicción de la configuración de disolventes en el descubrimiento de fármacos. Preimpresión disponible aquí: https://arxiv.org/abs/2309.12129.
Henriet, L. y col. (2020). Computación cuántica con átomos neutros. Cuántico, 4, 327.
Samways, ML y cols. (2021). Moléculas de agua en las interfaces proteína-fármaco: métodos de predicción y análisis computacional. Química. Soc. Rev., 50(16), 9104–9120.
Bucher, D. y otros (2018). Arrojando luz sobre aguas importantes para el diseño de fármacos: simulaciones versus métodos basados en redes. J. química. inf. Modelo., 58(3), 692–699.
Wlodawer, A. et al. (2008). Cristalografía de proteínas para no cristalógrafos, o cómo obtener lo mejor (pero no lo mejor) de las estructuras macromoleculares publicadas. FEBS J., 275(1), 1–21.
Michel, J. y otros (2009). Predicción del contenido de agua en los sitios de unión a proteínas. J. Física. Química. B,113(40), 13337–13346.
Barón, R. y otros (2010). Reconocimiento de agua en la cavidad-ligando. Mermelada. Química. Soc., 132(34), 12091–12097.
Fusani, L. y otros (2018). Redes de agua óptimas en cavidades proteicas con GAsol y 3D-RISM. Bioinformática, 34(11), 1947–1948.
Todo el proyecto de poner agua dentro de las proteínas será financiado por el programa Quantum for Bio. Lanzado en la primavera de 2023 por Wellcome Leap, sucesor de Wellcome Trust, el programa Quantum for Bio tiene como objetivo acelerar el uso de la computación cuántica en la atención médica mediante el desarrollo de aplicaciones que se beneficiarán de la llegada de las computadoras cuánticas dentro de 3 a 5 años..
¿Le gustaría aprender más sobre estas técnicas en una computadora cuántica de átomo neutro? Familiarícese con la computación cuántica, nuestra plataforma y los algoritmos con Quantum Discovery.



