Descubriendo patógenos: la evolución de la inmunidad vegetal

Al igual que los animales, las plantas tienen respuestas inmunitarias que les ayudan a defenderse de patógenos como virus, bacterias, hongos y oomicetos. Antes de poder detener a los invasores, primero deben detectarse, y esto se logra mediante receptores de reconocimiento de patrones ubicados en la superficie de las células vegetales. La capacidad de estos receptores para detectar patrones moleculares asociados con patógenos depende de dos tipos de proteínas, llamadas RLP y RLK, las cuales pueden contener repeticiones ricas en leucina: secciones que contienen el aminoácido leucina aparece varias veces.

Los receptores de inmunidad de las plantas detectan pequeños patógenos, como bacterias, una vez que ingresan. No pueden proteger contra la comida de las plantas.
Para rastrear la evolución de la inmunidad de las plantas, el equipo de investigación internacional dirigido por Ken Shirasu y Yasuhiro Kadota de RIKEN CSRS examinó la cantidad y los patrones de los receptores. Analizaron más de 170.000 genes que codifican RLK y alrededor de 40.000 genes que codifican RLP, que obtuvieron a partir de datos disponibles públicamente tomados de 350 especies de plantas. Descubrieron que los RLK y RLP con repeticiones ricas en leucina eran los tipos de receptores más abundantes entre todas las especies de plantas, representando casi la mitad de los RLK y el 70% de los RLP.
Se sabe que los RLP y algunos RLK contienen una región insular especial que es crucial para reconocer partes de los patógenos. Una investigación realizada por el equipo de RIKEN CSRS reveló que entre las RLP que contienen repeticiones ricas en leucina, esta región especial casi siempre estaba ubicada en el mismo lugar; entre la cuarta y quinta repetición rica en leucina. Se ha descubierto que estos RLP están asociados con respuestas inmunes. También descubrieron que la región insular estaba ubicada en la misma posición en algunos RLK, que casi todos pertenecen a un grupo funcional que regula el crecimiento y el desarrollo.
El análisis comparativo mostró que la secuencia de las cuatro repeticiones en la región de la isla era muy similar entre los dos tipos de detectores de proteínas, lo que sugiere que tienen un ancestro evolutivo común. En particular, estos cuatro conjuntos de repeticiones de leucina contenían secciones necesarias para unirse al mismo correceptor, llamado BAK1. Esto significa que las RLP relacionadas con el sistema inmunológico y las RLK relacionadas con el crecimiento heredaron la capacidad de unirse a BAK1 de un ancestro común.
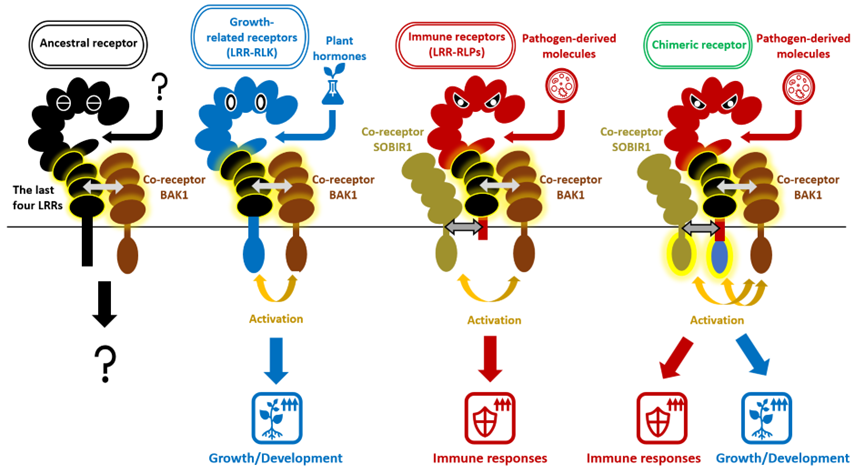
Tanto los LRR-RLP relacionados con el sistema inmunológico como los LRR-RLK relacionados con el crecimiento evolucionaron a partir de un ancestro común para heredar los últimos cuatro LRR y la capacidad de unirse al correceptor BAK1. El receptor quimérico que incorpora el dominio quinasa citoplasmático de un LRR-RLK relacionado con el crecimiento en un LRR-RLP relacionado con el sistema inmunológico activa tanto las respuestas inmunes como las de crecimiento cuando detecta una molécula derivada de un patógeno. LRR, repetición rica en leucina; RLP, proteína similar a receptor; RLK, quinasa similar a receptor.
«Curiosamente, descubrimos que el intercambio de las cuatro regiones de repeticiones ricas en leucina entre estos receptores no destruyó su funcionalidad», afirma Bruno Pok Man Ngou, quien llevó a cabo el estudio. La creación de un receptor híbrido combinando un RLK relacionado con el crecimiento con un RLP relacionado con la inmunidad dio como resultado un receptor híbrido que reconocía patógenos e inducía respuestas tanto inmunes como relacionadas con el crecimiento. Esto significa que los científicos deberían poder diseñar receptores con nuevas funciones intercambiando esos módulos.
Este estudio abordó el origen de la inmunidad vegetal a nivel molecular y demostró que al mismo tiempo el análisis de información de múltiples genomas vegetales puede permitir una predicción simple y precisa de los genes involucrados en la inmunidad y el crecimiento de las plantas. «Actualmente estamos aislando receptores inmunes de varias plantas utilizando esta información, con el objetivo de aplicaciones prácticas como el desarrollo de cultivos resistentes a enfermedades en el futuro», dice Shirasu.



